来源:病毒学界
2024 年 8 月 26 日,CRISPR 基因编辑先驱,2020 年诺贝尔化学奖得主 Jennifer A. Doudna 教授在 Nature 期刊发表了题为 Birth of protein folds and functions in the virome 的研究论文。该研究利用 AlphaFold 等 AI 工具预测了近 7 万个病毒蛋白质的 3D 形状,然后将新预测的结构与功能已知的蛋白质结构进行了匹配,为蛋白质的具体作用提供了新见解。研究还揭示了感染真核生物的病毒和感染细菌的病毒用于同一种进化上保守的古老的免疫逃逸机制,这些发现有助于开发新的抗病毒疗法。

图源:Nature
Jennifer A. Doudna 教授:随着具有大流行潜力的病毒的出现,确定它们将如何与人类细胞相互作用非常重要。这项新研究提供了一个工具来预测那些新出现的病毒可能带来的影响。
为了确定病毒蛋白质的功能,研究人员通常比较其独特的氨基酸序列与已知功能的蛋白质相似性。然而,由于病毒进化速度迅速,很多病毒蛋白质与已知蛋白质相似性不高。 研究团队使用 AlphaFold 预测蛋白质的三维结构,发现不同氨基酸序列的蛋白质可能具有相似的 3d 形状和功能。他们预测了来自近 4,463 种感染真核生物的病毒的 67,715 种蛋白质的结构,并通过深度学习工具对这些结构进行比对和分析。
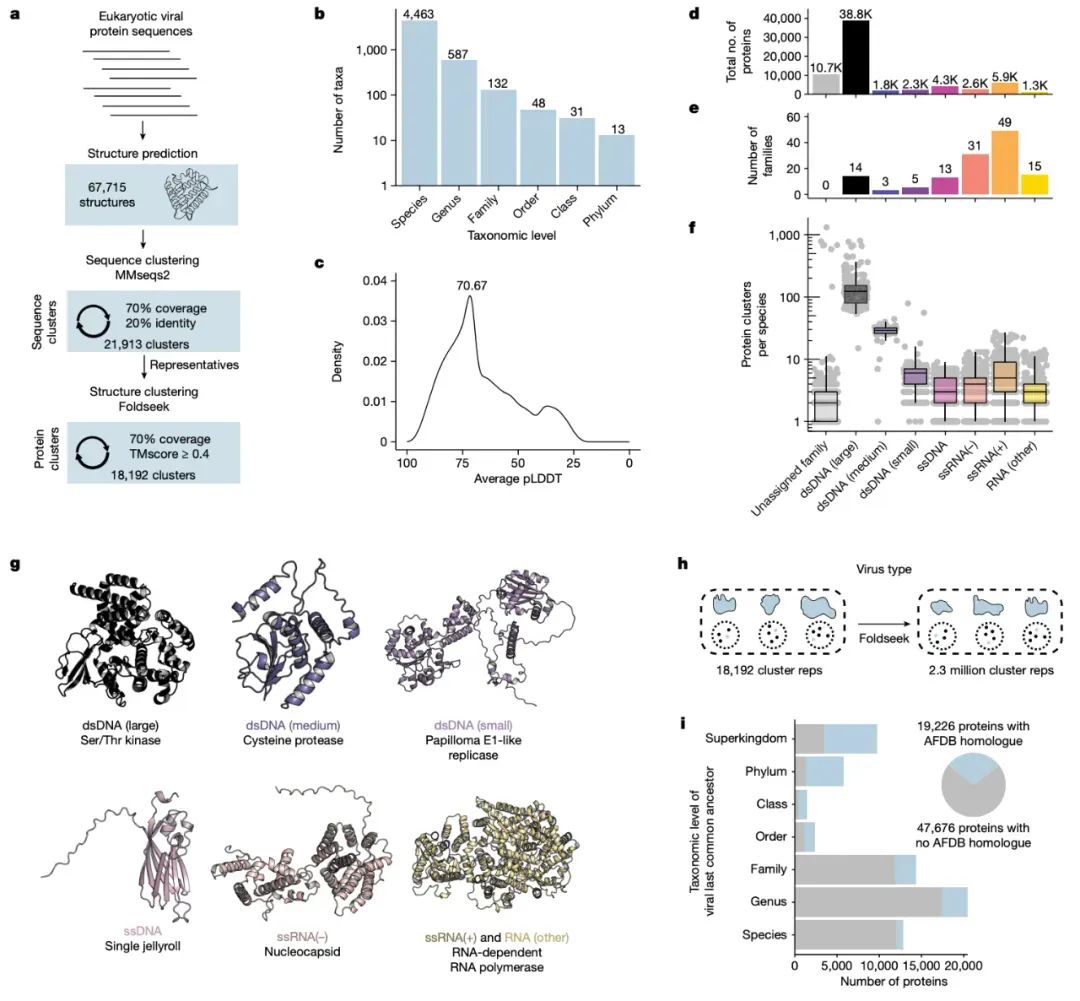
感染真核生物病毒的结构蛋白质组
Jason Nomburg 博士:如果没有计算工具取得的进步,我们无法准确且迅速地预测和比较蛋白质结构。研究团队发现,67,715 种新预测的病毒蛋白质中,62% 在结构上是独特的,缺乏同源物,而 38% 与已知蛋白质匹配,揭示了病毒与宿主之间的意外相似性。例如,一些新预测的病毒蛋白与哺乳动物细胞中用于转运 DNA 和 RNA 的非病毒蛋白惊人相似,暗示这些病毒蛋白可能具有转运功能。
进一步分析显示,多达 25% 的未注释病毒蛋白可能参与免疫逃逸,其中 RNA 连接酶 T 样磷酸二酯酶在感染真核生物的病毒与噬菌体中都广泛存在,表明这是一种广泛保守的免疫逃逸策略。从应对疾病的角度来看,突出了设计广泛有效的抗病毒疗法的新可能途径,例如,找到病毒免疫逃逸的常见、保守方式,可能会产生能同时有效对抗多种不同病毒的强效抗病毒药物。
本文及图片来源于病毒学界,作者转载