近年来,单细胞与空间组学领域文章呈井喷式增长。据 PubMed 数据库显示,仅 2023 年这一年里,单细胞/空间组学文章达一万多篇,其中位于 Q1 区,IF > 10 的文章突破 1,300 多篇。单细胞测序研究更是一路高涨,达 904 篇。
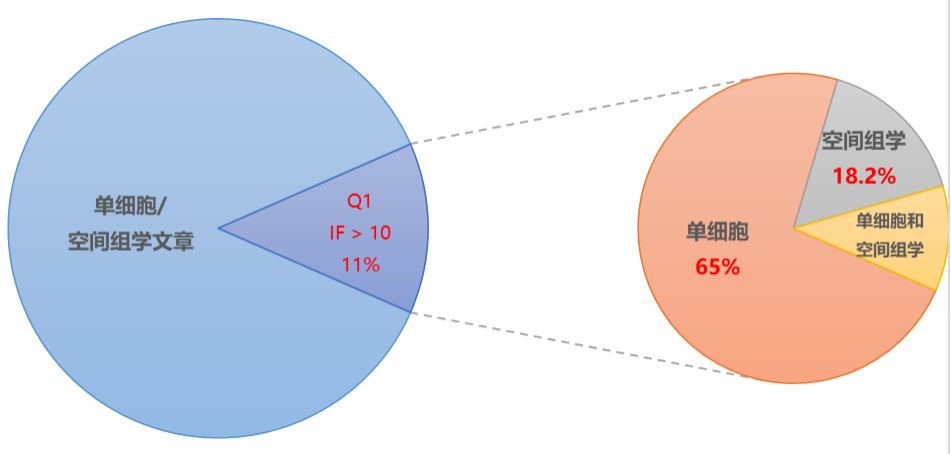
2023 年单细胞与空间组学文章统计
单细胞与空间转录组测序已应用于生物医学研究的各个领域。其中,涌现出不少里程碑式经典文章,我们盘点 2023 年那些值得复刻的经典文献!
1)人类细胞图谱计划
3 月份,剑桥大学 Roser 团队等绘制「迷你胎盘」图谱在 Nature 发表,为人类妊娠和早期胚胎发育提供了新的理论[1]。随后,胚胎肢体发育研究 A human embryonic limb cell atlas resolved in space and time 深入揭示了人类胚胎肢体在时间和空间上的发育特征,为先天性疾病的诊断提供理论依据[2]。此外,Nature 相继发布最全单细胞和空间分辨率心脏、乳腺、肠道、大脑以及肺部细胞图谱[3-5]。从早期妊娠、胚胎发育到器官细胞图谱,单细胞和空间组学技术一步步完善人类对复杂机体结构的理解,为人类疾病研究和药物开发开辟新途径。
2)组学新技术
6 月份,Science 发表研究 Linking genome structures to functions by simultaneous single-cell Hi-C and RNA-seq,国内邢栋团队开发 HiRES 体系,首次基于测序方法实现了在单细胞水平对转录组和三维基因组的同时检测,揭示三维基因组与基因表达的关系[6]。几乎同期,新型空间翻译组测序技术在 Science 发表,用于研究完整细胞和组织的 RNA调控和蛋白质合成[7]。9 月份,Cell 发表一项单细胞组学新方法— TrackerSci,揭示了大脑中不同祖细胞类型及表观遗传特征[8]。
3)癌症与免疫领域
8 月份,国内张泽民团队在 Cell 发文,通过大规模整合生物信息分析揭示泛癌组织中 NK 细胞异质性[9]。Li Ding 团队在 Nature 发表研究成果,成功绘制 11 种癌症的多组学图谱,揭开癌症发展过程中的表观遗传调控机制[10]。在肿瘤转移方面,Peter M. Siegel 团队等人绘制脑肿瘤空间免疫图谱揭示了原发肿瘤和不同实体癌脑转移之间免疫景观的差异,以及胶质母细胞瘤患者生存相关的细胞邻区[11]。
4)脑科学与神经系统疾病
Cell 发表研究提出最为全面的人脑多区域时空发育图谱及规律,为人脑发育的区域性研究提供新见解[12]。Andrea Volterra 等团队更是发现一种新型脑细胞—谷氨酸能星形胶质细胞,提示星形胶质细胞在中枢神经系统疾病治疗中的潜在靶点,相关研究 Specialized astrocytes mediate glutamatergic gliotransmission in the CNS 在 Nature 发表[13]。众所周知,淀粉样蛋白沉积是阿尔兹海默症(AD)的重要病理标志,Nature 发表研究指出髓磷脂功能障碍和脱髓鞘损伤是蛋白沉积的驱动力,改善髓鞘健康可延缓 AD 发病[14]。
左右滑动查看更多

单细胞与空间技术新突破
AGBT(基因组生物学技术进展)大会作为全球基因组学和测序技术领域规格最高的年度会议,为全球顶级研究者提供交流和技术共享的平台。今年 AGBT 不仅带来了组学领域最新进展,还发布了单细胞与空间技术新突破。
10x Genomics 发布三个平台新技术—Visium 的高清晰度助力实现空间探索,Xenium 的超高精度推动单细胞空间分析,Chromium 的多功能性推动单细胞领域研究兼具深度与广度。
令人翘首期盼的高分辨率-提升 Visium 的空间发现能力
Visium 空间平台帮助研究人员在空间背景下进行无偏倚的全转录组分析。FFPE 兼容性更是有利于临床样本的病理研究。随着研究的深入,对空间分辨率也提出了更高的要求。为此,我们将进一步提升分辨率,实现高分辨率下的全转录组空间分析。
➤ Visium HD 的优势
1
重新设计的捕获区域为单细胞分辨率赋能
捕获区域密布有连续的带有条形码的寡核苷酸,排列在数百万个没有间隙的正方形中,而 CytAssist 促进了精准的转录本定位。
2
与人类和小鼠的多种 FFPE 组织兼容
采用几乎通用的样本制备方案,这意味着大多数组织都无需优化。
3
Visium v2 FFPE 化学试剂
该试剂所使用的基于探针的高灵敏度基因表达检测方法让 HD 可达到相当甚至略高的灵敏度。
4
同一张切片的 H&E 和 IF 整合
能够将基因表达数据与形态特征相关联,方便进行正交验证并提高可信度。
➤ 示例—Visium HD 如何在高分辨率下保持空间性
这是小鼠肠道,现在展示的是隐窝,隐窝内是潘氏细胞(paneth cells)。利用在分子数据上叠加 H&E 图像的功能,手动标注包含这些潘氏细胞的区域,然后查看 Lyz1 和 Defa21 这两个基因。预计只会出现在潘氏细胞上,这一点确实在图中得到了证实。虽然只是展示了一两个基因,但这是从每个实验中近 2 万个基因的数据中提取出来的。
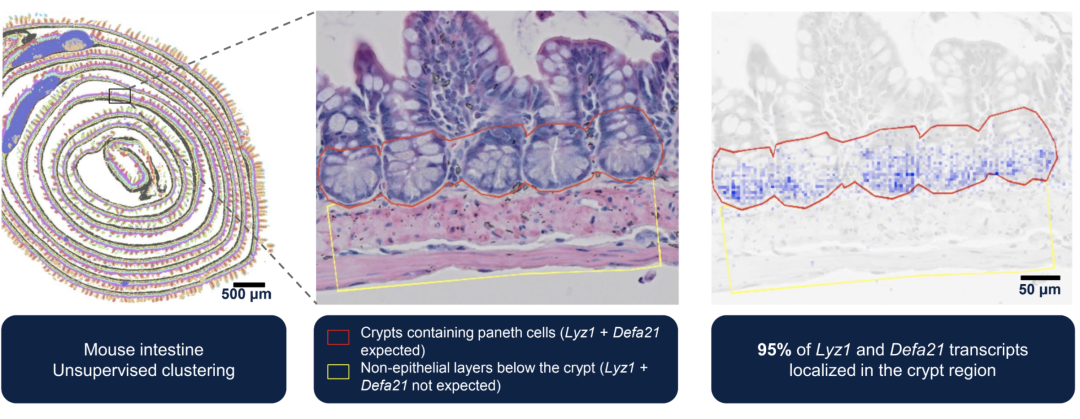
上图展示了 Visium HD 能够保持空间性,并在小鼠肠道隐窝内的潘氏细胞的预期位置上显示经典标志物基因的位置。
扩展 Xenium 的功能—获得更强大的单细胞空间洞察力
借助 Xenium 原位分析平台,已经有不少文章发表。有别于传统分析方法,Xenium 平台进行了功能与算法优化,得到广大科研人员的一致好评。多模态细胞分割的功能是利用抗体混合物和严格训练的算法,根据细胞边界(一级)、内部(二级)或细胞核(三级)的染色结果对细胞进行分割。这种方法在人类和小鼠的多种 FFPE 组织中显示出良好的兼容性。这种新的多模态细胞分割也会整合到未来的 panel 中。
➤ 多模态分割方法的优势:
5,000 个基因的 panel 采用了全新的分子结构、光学条形码设计和算法堆栈,让每个基因都保持高灵敏度。
可在 6 天时间内对两张满载的载玻片上总面积达 4 cm2 的组织样本运行分析。
未来将推出多重的蛋白质分析解决方案,实现 RNA 和蛋白质的整合分析。
➤ 示例——多模态分割方法的重要性
图中高亮显示了一些成纤维细胞。由于边界有误,它们从旁边的胰岛细胞中获得了一些信号,并被错误标注出来。如果正确地分割细胞,就能看到拉长的形态,并获得正确的细胞类型分配。

GEM-X——开启单细胞分析新时代
Chromium 分析取得重大突破,与 FFPE 兼容的 Chromium Flex 分析将具备新的分析物检测功能,包括细胞内蛋白和 CRISPR。此外,Flex 能够在 scRNA-seq 前固定全血样本,让液体活检研究更加灵活方便。全新突破!全新的 Chromium GEM-X 单细胞基因表达 v4 和 GEM-X 单细胞免疫分析 v3 分析(现正接受预订)将采用 GEM-X 技术,并与 Chromium X/iX 仪器无缝整合。
➤ Chromium GEM-X 的优势:
• 检测到的基因数多达 2 倍
• 细胞通量提高 2 倍
• 每个细胞的成本降低 2 倍
• 多细胞比例降低 2 倍
• 细胞回收率高达 80%
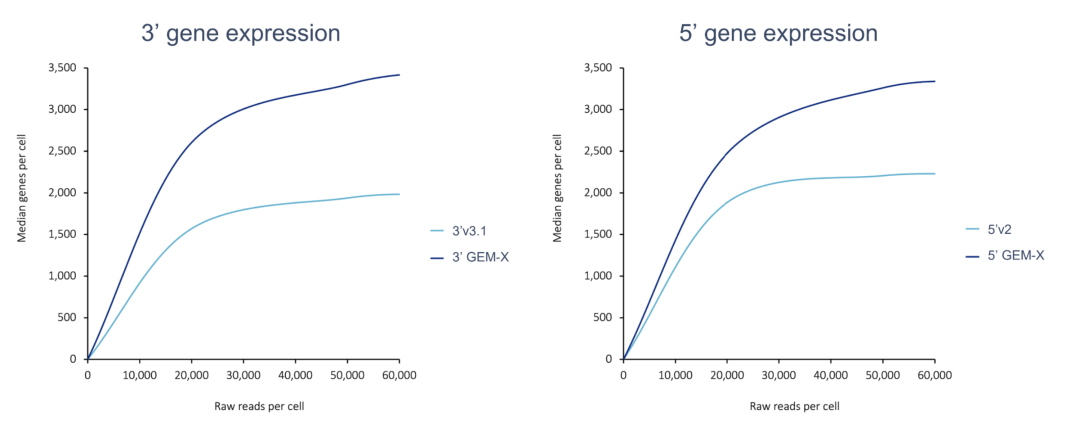
采用 Chromium GEM-X 分析来检测成年小鼠大脑细胞核这种众所周知的复杂样本类型时,基因表达灵敏度大大提高,最高达到 80%。
➤ 示例—GEM-X 如何处理复杂样本
在处理最复杂的样本时,GEM-也能获得最准确的结果。下面的数据显示了 GEM-X 如何捕获中性粒细胞。因为它们的 RNA 含量很低,这种细胞类型一直都是单细胞分析的难点。

从 Chromium GEM-X 单细胞基因表达 v4 分析(3’ GEM-X)中收集的数据显示,中性粒细胞(深紫色细胞簇)的检测能力增强。
对于 5’ 免疫分析试剂盒,GEM-X 的优势在于能够获得更多 V(D)J 信息,包括利用丰富的 V(D)J 转录本揭示更多的独特克隆型。
更多研究进展和技术创新未完待续……
立即联系 10x Genomics
询价或了解更多信息

内容策划:潘成
内容审核:朱超敏
题图来源:图虫创意
图片来源:10x Genomics
参考文献:
[1]. Arutyunyan A, Roberts K, Troulé K, et al. Spatial multiomics map of trophoblast development in early pregnancy. Nature. 2023 Apr;616(7955):143-151. doi: 10.1038/s41586-023-05869-0.
[2]. Zhang B, He P, Lawrence JEG, et al. A human embryonic limb cell atlas resolved in space and time. Nature. 2023 Dec 6. doi: 10.1038/s41586-023-06806-x.
[3]. Kanemaru K, Cranley J, Muraro D, et al. Spatially resolved multiomics of human cardiac niches. Nature. 2023 Jul;619(7971):801-810. doi: 10.1038/s41586-023-06311-1.
[4]. Kumar T, Nee K, Wei R, et al. A spatially resolved single-cell genomic atlas of the adult human breast. Nature. 2023 Aug;620(7972):181-191. doi: 10.1038/s41586-023-06252-9.
[5]. Sikkema L, Ramírez-Suástegui C, Strobl DC, et al. An integrated cell atlas of the lung in health and disease. Nat Med. 2023 Jun;29(6):1563-1577. doi: 10.1038/s41591-023-02327
[6]. Liu Z, Chen Y, Xia Q, et al. Linking genome structures to functions by simultaneous single-cell Hi-C and RNA-seq. Science. 2023 Jun 9;380(6649):1070-1076. doi: 10.1126/science.adg3797.
[7]. Zeng H, Huang J, Ren J, et al. Spatially resolved single-cell translatomics at molecular resolution. Science. 2023 Jun 30;380(6652):eadd3067. doi: 10.1126/science.add3067.
[8]. Lu Z, Zhang M, Lee J, et al. Tracking cell-type-specific temporal dynamics in human and mouse brains. Cell. 2023 Sep 28;186(20):4345-4364.e24. doi: 10.1016/j.cell.2023.08.042.
[9]. Tang F, Li J, Qi L, et al. A pan-cancer single-cell panorama of human natural killer cells. Cell. 2023 Sep 14;186(19):4235-4251.e20. doi: 10.1016/j.cell.2023.07.034.
[10]. Terekhanova NV, Karpova A, Liang WW, et al. Epigenetic regulation during cancer transitions across 11 tumour types. Nature. 2023 Nov;623(7986):432-441. doi: 10.1038/s41586-023-06682-5.
[11]. Karimi E, Yu MW, Maritan SM, et al. Single-cell spatial immune landscapes of primary and metastatic brain tumours. Nature. 2023 Feb;614(7948):555-563. doi: 10.1038/s41586-022-05680-3.
[12]. Li Y, Li Z, Wang C, et al. Spatiotemporal transcriptome atlas reveals the regional specification of the developing human brain. Cell. 2023 Dec 21;186(26):5892-5909.e22. doi: 10.1016/j.cell.2023.11.016.
[13]. de Ceglia R, Ledonne A, Litvin DG, et al. Specialized astrocytes mediate glutamatergic gliotransmission in the CNS. Nature. 2023 Oct;622(7981):120-129. doi: 10.1038/s41586-023-06502-w.
[14]. Depp C, Sun T, Sasmita AO, et al. Myelin dysfunction drives amyloid-β deposition in models of Alzheimer's disease. Nature. 2023 Jun;618(7964):349-357. doi: 10.1038/s41586-023-06120-6.
点击「阅读原文」询价或了解更多信息