H&E 染色是对组织切片进行组织结构、疾病病理分析的经典方法,几乎是科研人员必备技能。但是如果想基于染色切片进行组学实验检测时,往往存在诸多技术瓶颈。
在实验过程中,H&E 染色通常需要丰富的病理学经验辅助判断组织结构,并且特定区域的全蛋白表达水平对疾病结果解读也至关重要!但对 H&E 染色切片进行蛋白质谱检测时,染色染料的干扰会降低质谱数据的准确性和稳定性,甚至导致仪器受损。基于此,传统的空间蛋白质组学采用临片检测的策略(即连续切片,一张切片进行染色,相邻切片对应位置进行质谱检测),但这又导致了「非原位」误差干扰。
为解决上述困境,贝普奥生物采用全新的 H&E 切片空间蛋白质组技术,可对同一张切片目标区域同时进行染色和蛋白质谱检测,有效提高 H&E 染色切片的空间蛋白质组学的精度和深度!(文末有福利)

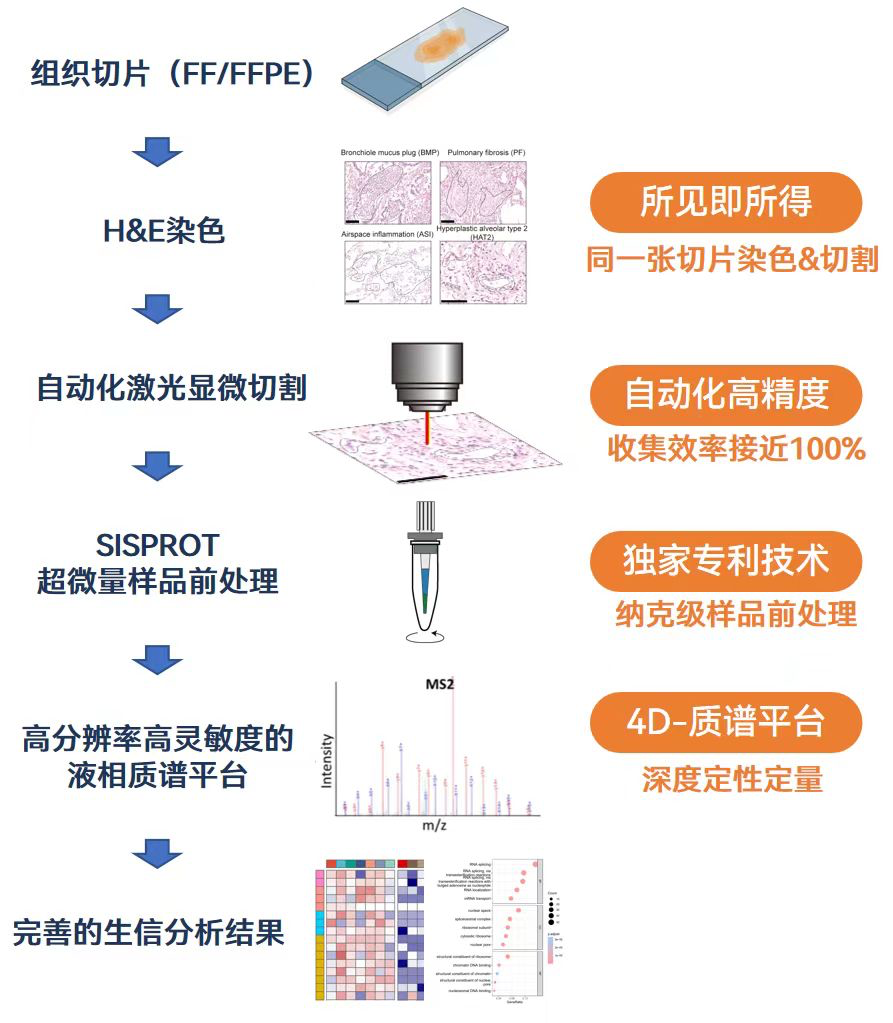
H&E 空间蛋白质组技术流程及优势
01.完整的空间蛋白质组学技术流程
获取组织切片(FF/FFPE)、H&E 染色、自动化激光显微切割、SISPROT 超微量样品前处理、高分辨率高灵敏度的液相质谱平台以及获得最终生信分析结果。
02.空间蛋白质组学技术特点
1
基于 H&E 染色,可在同一张切片上轻松实现组织微环境的高效准确区分、轮廓标注和激光显微切割。
2
自动化精准切割、精准定位和 100% 目标样本收集,保证了目标样本质谱检测的一致性,提升蛋白定量的准确性和稳定性。
3
完美实现显微切割样本的高通量、高深度蛋白质组鉴定,纳克级微量样本也可进行高效分析。

H&E 空间蛋白质组疾病研究应用
H&E 空间蛋白质组揭示肺损伤机制
前不久,BioRxiv 上线中科院武汉病毒研究所石正丽团队与南方科技大学田瑞军团队以及华中科技大学同济医学院周亦武团队的创新性研究 Deep spatial proteomic exploration of severe COVID-19-related pulmonary injury in post-mortem specimens,通过整合 H&E 组织切片染色、单细胞精度激光显微切割(LMD)和超灵敏空间蛋白质组学技术(SISPROT),对武汉大流行第一批死亡患者留存的 FFPE 样品进行高深度空间蛋白质组学检测,建立了 COVID-19 患者肺部七个主要病理区域的高分辨定量蛋白质组学图谱,在 71 个解剖的 FFPE 死后标本中定量分析了超过 10,000 种蛋白质。比较发现了 COVID-19 诱导的蛋白谱和信号通路失调,为过渡态肺细胞增殖提供了证据。
本研究为了解 COVID-19 引起的肺损伤提供了空间蛋白质组学信息,为重症肺炎的治疗干预提供重要理论依据[1]。
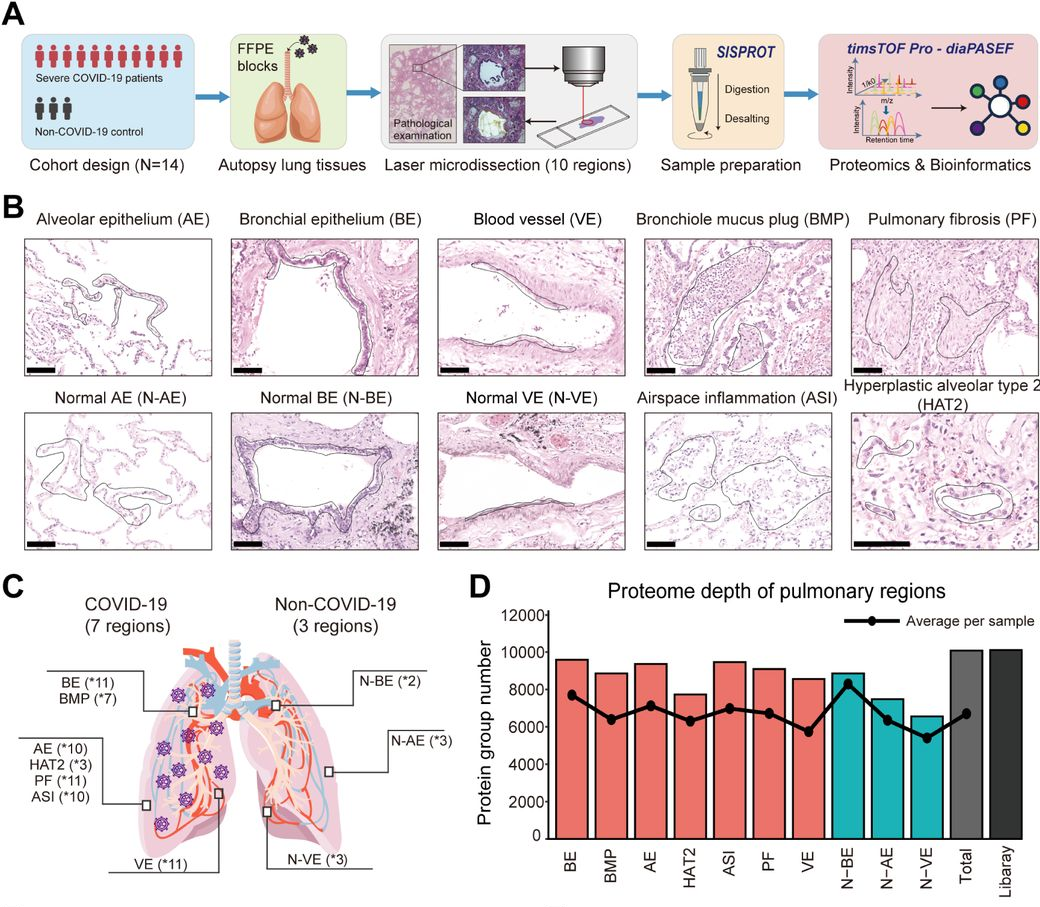
图 1. COVID-19 尸检中肺损伤的空间蛋白质组分析
A. 实验流程:队列设计、尸检标本采集、激光显微切割、蛋白质组学样品制备、基于质谱的深度蛋白质组学分析、生物信息学分析。对 COVID-19 重症患者和非 COVID-19 对照个体的肺组织十个区域进行分析。B. 组织切片 H&E 染色分析。C. 人体肺结构示意图。D. 每个区域已识别蛋白质组的总数(条形图)和平均值(折线图)。
(上下滑动查看)
H&E 空间蛋白质组揭示肿瘤组织异质性
早在 2018 年,南方科技大学田瑞军课题组利用 H&E 空间蛋白质组技术,在 Analytical Chemistry 上发表研究 Spatial-Resolution Cell Type Proteome Profiling of Cancer Tissue by Fully Integrated Proteomics Technology,研究基于 H&E 染色切片,结合激光显微解剖和蛋白质组学样品制备技术 SISPROT,在单细胞分辨率水平确定肿瘤样品中的特定细胞类型,并进行高灵敏度蛋白质组分析。首次获得了来自同一结肠肿瘤和周围正常组织的四种细胞类型(癌细胞、肠细胞、淋巴细胞和平滑肌细胞)的组织分布和空间维度的蛋白表达谱[2]。
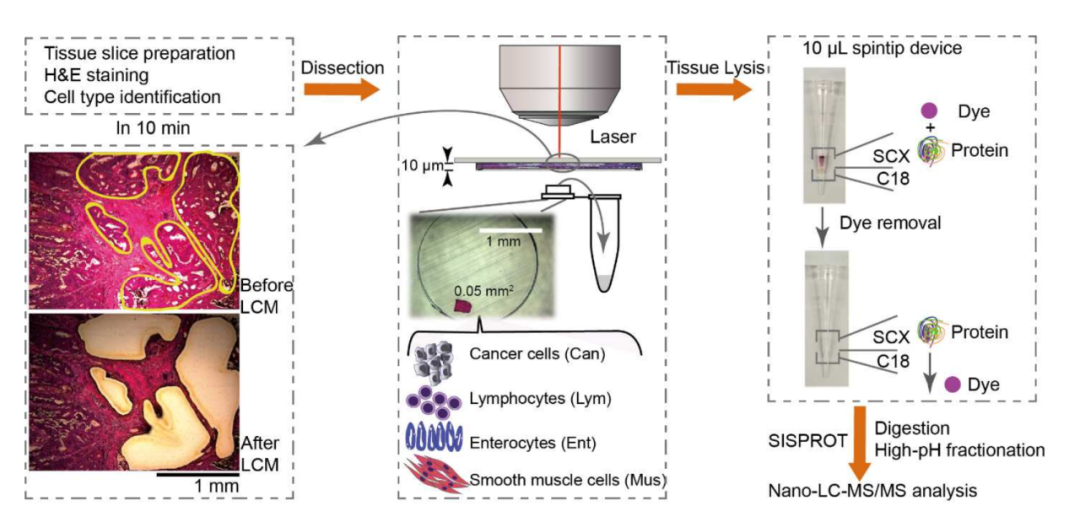
图 2. 结肠癌组织 H&E 空间原位蛋白质组分析流程图
(上下滑动查看)
年终岁末,为了给大家提供更好的空间蛋白质组学技术支持,贝普奥生物推出 2 周年钜惠活动,H&E 空间蛋白质组限时超值优惠、10 天极速交付结果,做空间蛋白质组学的你千万别错过!(详见下方海报)

内容策划:潘成
内容审核:周育红
图片来源:贝普奥生物
题图来源:图虫创意
参考文献
[1] Yiheng Mao, Ying Chen, Yuan Li, et al., Deep spatial proteomic exploration of severe COVID-19-related pulmonary injury in post-mortem specimens, bioRxiv, 548971, 2023.
[2] Xu R, Tang J, Deng Q, et al., Spatial-Resolution Cell Type Proteome Profiling of Cancer Tissue by Fully Integrated Proteomics Technology, Anal. Chem., 2018, 90(9): 5879-5886.