2023 年 1 月 12 日,哈佛医学院 David A. Sinclair 团队在核心期刊 Cell 上发表一篇题为 Loss of epigenetic information as a cause of mammalian aging 的论文引起学术界的广泛关注[1]。
经过长达 13 年研究,阐述了 DNA 变化不是导致衰老的唯一原因。相反,他们的研究结果表明,染色质的化学和结构变化在不改变遗传密码本身的情况下加快衰老。即所谓的表观遗传学的退化可以驱动有机体的衰老,恢复表观基因组的完整性可以逆转衰老的迹象,而与遗传密码本身的变化无关。
那么这些表观遗传因子是如何参与发生衰老与逆转呢?这就不得不提到表观遗传学的物理结构,如组蛋白,它能将 DNA 捆绑成紧密压缩的染色质,并在需要时解开 DNA 的部分,用于调控基因表达或抑制,这一研究称之为染色质可及性研究。
什么是染色质可接近性/开放性?
DNA 与组蛋白结合后形成核小体,核小体再进一步折叠压缩后最终形成染色质。DNA 复制、基因转录时,DNA 的致密高级结构变为松散状态,这部分打开的染色质,称之为开放染色质。
染色质一旦打开,就能让某些调节蛋白,例如转录因子和辅因子与其结合,这种染色质的性质,被称为染色质可接近性[2]。该性质是表征染色质转录活性的重要指标,也被称作「研究基因功能的窗口」。在特殊情况下,染色质开放度的改变能够为我们揭示基因表达的调控机制提供重要信息。
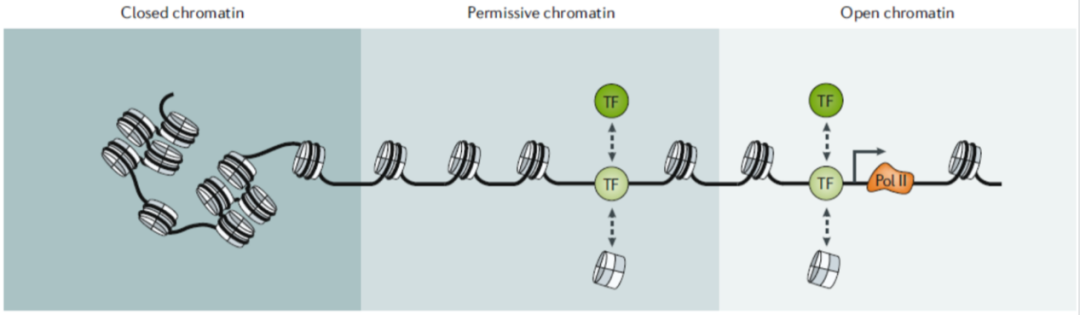
图 1. 可接近状态反映基因组中染色质动态的分布
注:与封闭染色质(closed chromatin)相反,宽松染色质(permissive chromatin)对于转录因子来说是足够动态的,可以启动序列特异性的可及性重塑并建立开放染色质(open chromatin)构象。
什么是 ATAC-seq?
ATAC-seq(Assay for Transposase-Accessible Chromatin using sequencing)是利用转座酶研究染色质可及性的高通量测序技术。
美国斯坦福大学 William Greenleaf 教授于 2013 年研究了一种基于 Tn5 酶对染色质解离的新技术,该技术的基本原理是通过对 Tn5 酶所捕获的 DNA 序列进行测序,获得目标 DNA 序列信息[3]。转座酶能对染色质开放区中的 DNA 进行随意的结合和剪切,并能在剪切位点上插入连接序列。与 MNase-seq、DNase-seq 等方法相比,该方法具有操作简单、检测周期短、需要的细胞/组织数量少、检测结果质量好等优点。
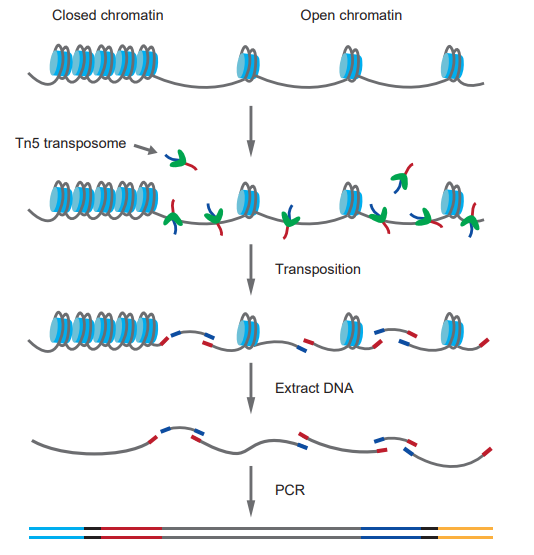
图 2. ATAC-seq 原理与实验步骤
ATAC-seq 的应用场景有哪些?
”
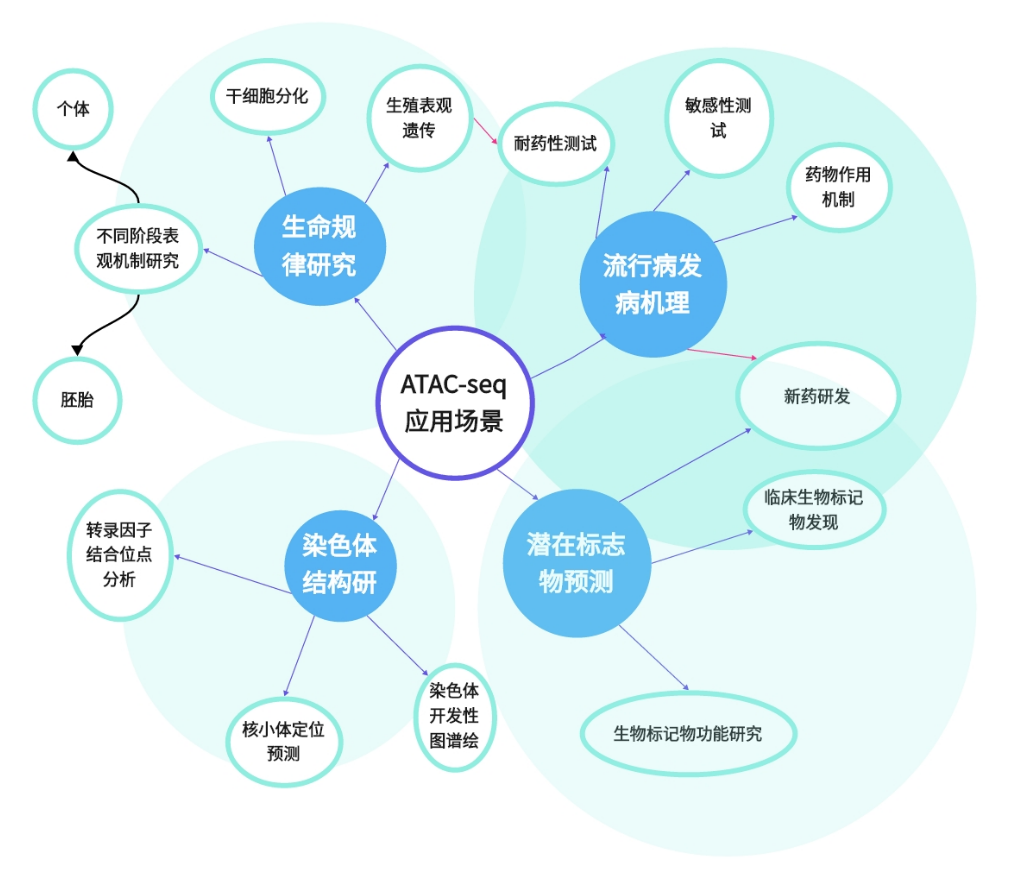
ATAC-seq 案例分享
”
1.
免疫研究
该工作通过单细胞 ATAC-seq 技术以及新开发的单细胞多组学 Ti-ATAC-seq 技术,描绘了新冠肺炎康复者外周免疫细胞可接近染色质多维图谱,解析了康复者中单核细胞、B 细胞和 T 细胞免疫记忆形成的表观遗传机制[4]。该研究结果表明,新冠肺炎康复者的天然免疫细胞和适应性免疫细胞都发生了大量的染色质重塑。康复者外周血中富集了 TBET-enriched CD16+ 和 IRF1-enriched CD14+ 单核细胞,经确定具有训练有素(trained)和激活的表观状态。
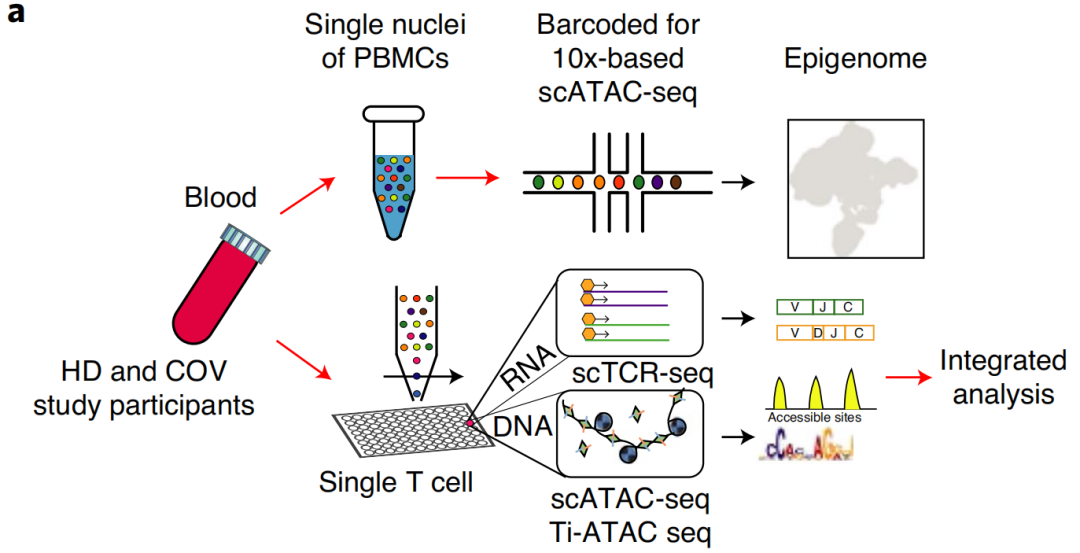
图 3. 单细胞多组学 Ti-ATAC-seq 技术
2.
胚胎发育
该研究采用 ATAC-seq 技术揭示人类胚胎合子基因组激活(ZGA)前存在广泛的染色质开放区域,并阐明其在胚胎发育过程的重编程模式,阐述了胚胎基因组转录激活对于开放染色质区域重编程的必要性[5]。该研究团队优化并建立了可识别低至 50 个细胞中的组蛋白修饰染色质定向捕获分割技术(CUT & RUN),并针对人类早期胚胎样本进行了优化,进一步在人类卵母细胞、合子基因组激活(ZGA)前后胚胎和内细胞团等时期检测了 H3K4me3 和 H3K27me3 的动态变化,在 ZGA 后的胚胎中检测了 H3K27ac 的分布:研究发现人类早期胚胎发育过程中的组蛋白重编程经历了和小鼠不同的动态变化。
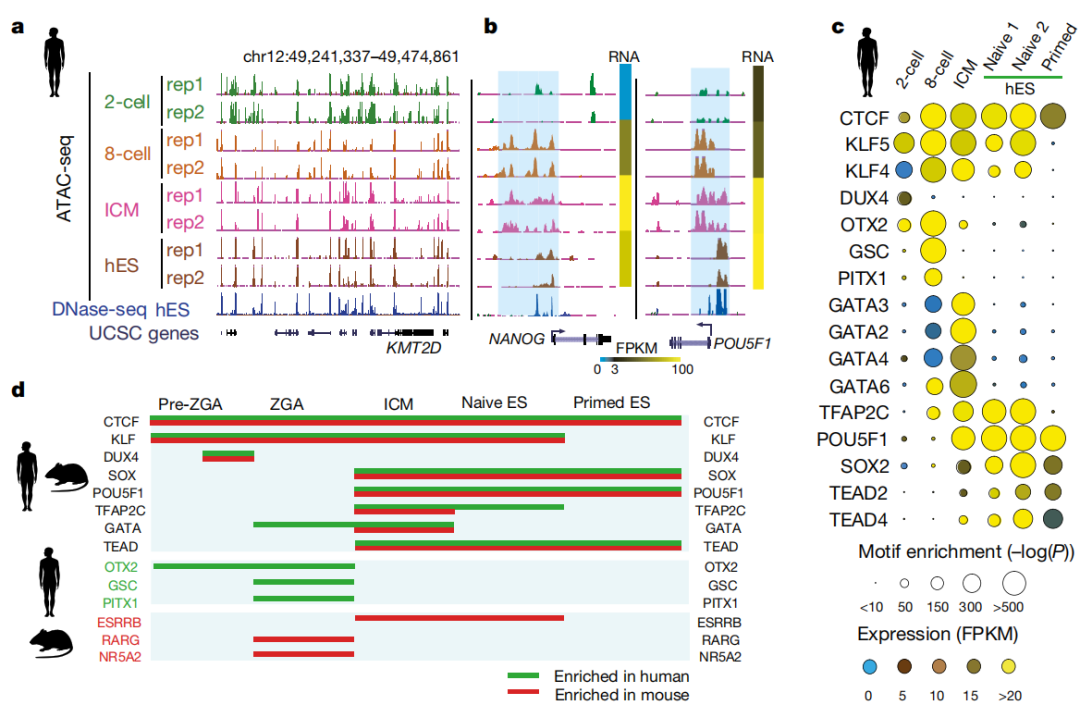
图 4. 人类早期胚胎染色体上存在大量的开放区域(从 2 细胞到 8 细胞期)
(上下滑动查看)
ATAC-Seq 为表观遗传学研究提供了更快捷有效的方式,越来越多的研究者们应用 ATAC-seq 技术,在肿瘤、免疫、发育等领域获得了突破性研究。
如何入门 ATAC-Seq?科研人必冲的实战训练营来啦!从技术讲座到实操带教,手把手教你搞定 DNA-蛋白质互作研究
▼ 扫码解锁行程安排 ▼

诺唯赞关联产品速递
”
内容策划:潘成
内容审核:吴军
题图来源:视觉中国
参考文献
[1] Jae-Hyun Yang et al. Loss of epigenetic information as a cause of mammalian aging. Cell, 2023, doi:10.1016/j.cell.2022.12.027.
[2] Klemm, S.L., Shipony, Z. & Greenleaf, W.J. Chromatin accessibility and the regulatory epigenome. Nat Rev Genet 20, 207–220 (2019)
[3] William J.Greenleaf, Jason D. Buenrostro, et al. Transposition of native chromatin for multimodal regulatory analysis and personal epigenomics. Nat Methods. 2013, 10(12): 1213–1218
[4] Schwartzman, O., Tanay, A. Single-cell epigenomics: techniques and emerging applications. Nat Rev Genet 16, 716–726 (2015)
[5] Wu, J., Xu, J., Liu, B. et al. Chromatin analysis in human early development reveals epigenetic transition during ZGA. Nature 557, 256–260 (2018)
 点击「阅读原文」,在线咨询
点击「阅读原文」,在线咨询