表观遗传学的研究离不开对蛋白质-DNA 相互作用的探究,其中不得不提的就是独特技术原理与高通量完美结合的 ChIP(Chromatin Immunoprecipitation Assay)技术,但在使用过程中却有很多问题对新手小白非常不友好,让他们流尽辛酸泪(文末含惊喜彩蛋  ):
):
➣ 细胞起始量要求高(> 10^6)
➣ 交联不当易出现假阴/阳性
➣ 超声打断条件需要大量经验
➣ 高度依赖特异性抗体
➣ 重复性差、信噪比低
➣ 实验周期长(3~4 天)
……
因此,更加方便省时的 ChIC 和 ChEC 技术应运而生,但这个技术只能通过 Southern blot 对特定位点进行分析,无法满足高通量测序时代的需求,渐渐无人问津。
直到它的出现,可以帮助科研人摆脱「折磨」的 ChIP 实验,使蛋白质-DNA 相互作用研究变得更便捷、高效、准确。那就是 Henikoff 团队发布的 CUT&RUN 和 CUT&Tag 技术,不论是从操作难度或是数据质量上看,CUT&RUN 、 CUT&Tag 与 ChIP 技术相比都略胜一筹(图 1)。

图 1 ChIP 与 CUT&RUN、CUT&Tag 的比较
一
ChIP 的革新性技术-CUT&RUN
CUT&RUN 技术由 Henikoff 团队于 2017 年首次在 Elife 上报道[1],并在后续进行了数次改进,最终获得了较为成熟的 CUT&RUN 技术。
该技术的革新点在于使用 ConA beads 通过膜表面糖蛋白结合细胞,并用洋地黄皂苷(Digitonin)对细胞膜打孔,使抗体和 pA-MNase 进入活细胞并对目的位点进行切割(图 2)。在活细胞中进行靶向切割,可以避免甲醛交联造成的蛋白质的交联、抗原表位的掩盖和大量细胞损失,降低假阴性和假阳性的概率。
与 ChIP 相比,CUT&RUN 的优点不仅仅体现在实验流程的简化,更体现在性能的提升,如转录因子成功率高、抗体兼容性强、富集效果好以及信噪比高等(图 3)。
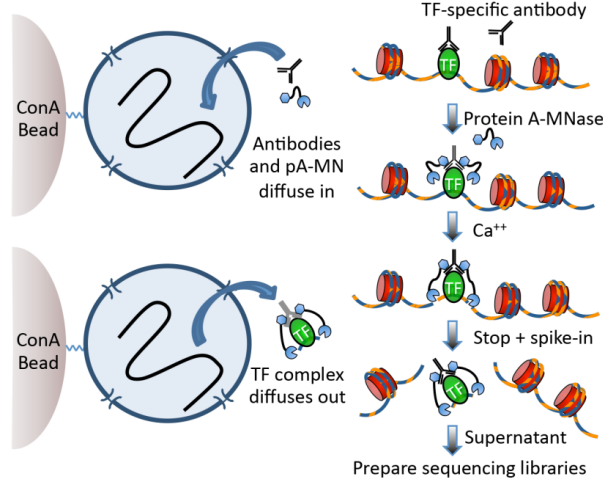
图 2 CUT&RUN 技术原理[1]
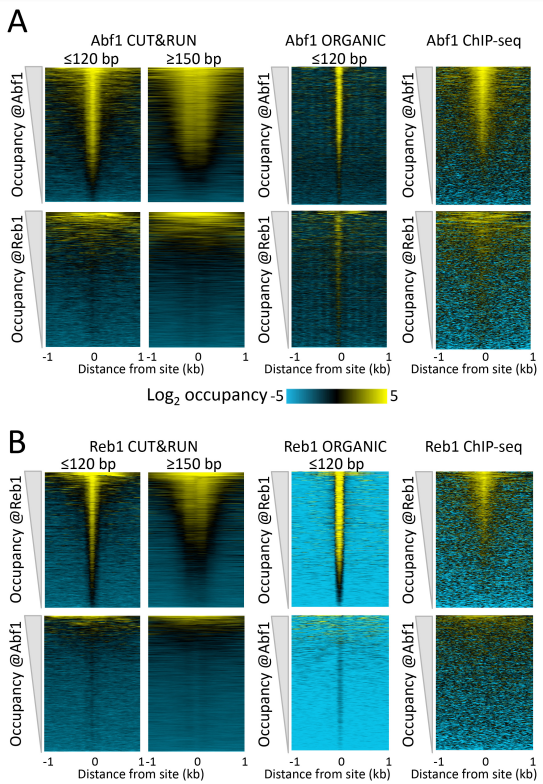
图 3 CUT&RUN-seq 和 ChIP-seq TSS 富集效果的比较[1]
CUT&RUN 技术在发育生物学、肿瘤发生机制等方面的研究都有着出色的发挥。Cheng-Ming Chiang 团队发现致癌 BRD4-S 和抑癌 BRD4-L 在乳腺癌的发生和发展中具有相反的功能,为乳腺癌的治疗提供了理论基础,研究者利用 CUT&RUN 技术和 ChIP 技术相互佐证,验证了 CUT&RUN 技术的可靠性和高质量[2](图 4)。
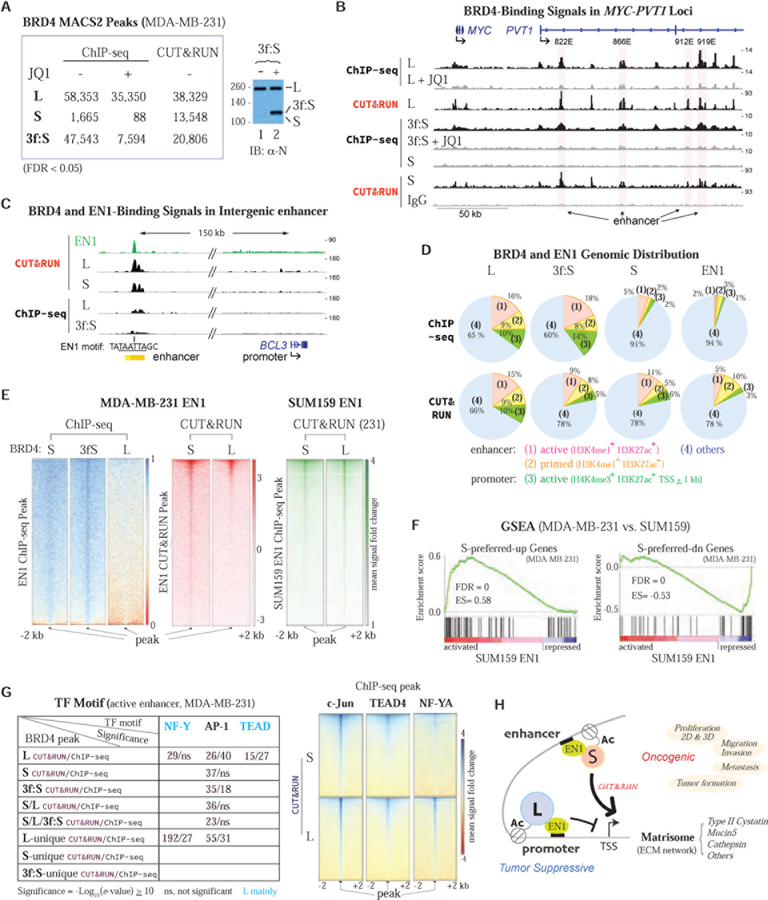
图 4 通过 ChIP-seq 和 CUT&RUN 研究 BRD4-S 和 EN1 的全基因组关联[2]
▲(上下滑动查看)▼
二
更少的细胞更好的兼容-CUT&Tag
虽然 CUT & RUN 技术已经基本解决了 ChIP 技术弊端带来的负面影响,但 CUT&RUN 有一个必需的 NGS 接头连接步骤,这就使其很难适应微量细胞的应用。
为了克服这种困难,Henikoff 团队于 2019 年 4 月在 Nature Communications 上首次公布了 CUT&Tag 技术[3],使用包埋了接头的转座酶 Tn5 来替代核酸酶 MNase,虽然 Tn5 对 DNA 的片段化效率不如 MNase,但 Tn5「粘贴」的特性可使其在切割目的位点的同时,在目的片段两端添加建库接头,大大简化了建库流程,兼容更低的起始细胞投入量,同时,以 Protein A/G 代替 Protein A 提高了对抗体的兼容性。

图 5 CUT&Tag 技术原理[3]
CUT&Tag 在疾病发生机理研究方向的贡献尤为突出。南京医科大学王晓明教授团队在类风湿性关节炎的发病机制的研究中,应用 CUT&Tag 技术分析了 H3K9me2 的全基因组沉积,发现 Jmjd1c 对 STAT3 磷酸化的调节是独立于组蛋白去甲基化的[4](图 6)。
不仅如此,CUT&Tag 的实验样本也从哺乳动物细胞样本扩展到植物样本,华中农业大学李林教授团队制备了水稻和玉米原生质体作为样本,以转录因子为靶蛋白进行 CUT&Tag 实验,并与 ChIP 进行平行对比[5](图7),结果表明了植物原生质体 CUT&Tag 的可行性,该实验的成功是 CUT&Tag 技术的突破和应用范围的进一步拓展。
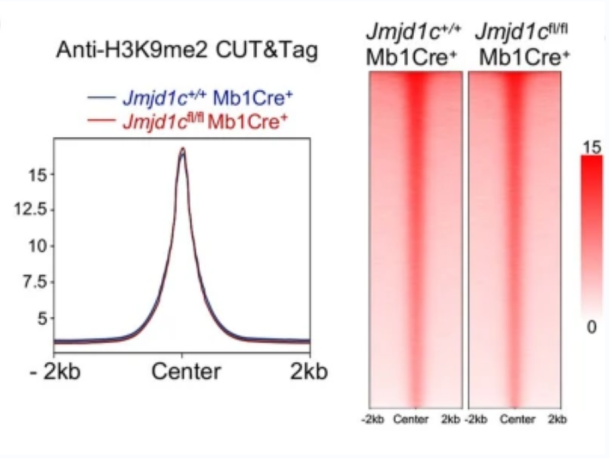
图 6 Jmjd1c 不调节 B 细胞中的 H3K9 甲基化[4]
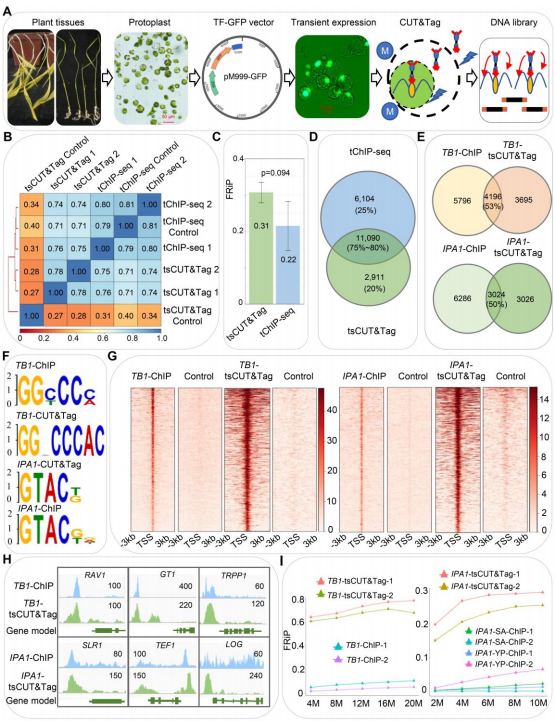
图 7 一种瞬时和简化的 CUT&Tag 方法分析植物中转录因子结合位点[5]
CUT&Tag 便捷高效,可兼容更低细胞起始量,但 CUT&RUN 也有自己更占优势的应用空间。与转座酶 Tn5 相比,核酸酶 MNase 的 DNA 片段化能力更强,能实现单碱基切割,在目的蛋白丰度较低的情况下也能高效捕获并切割目的位点,因此 CUT&RUN 对低丰度转录因子的实验效果更佳,这一点也得到了 Henikoff 的认同。由于 MNase 比 Tn5 的占位更小,对异染色质的切割更理想,对于 H3K9me3 这类位点 CUT&RUN 比 CUT&Tag 的实验效果更好。
因此,对 CUT&Tag 和 CUT&RUN 的选择是因「人」而异、因「地」制宜的,一个恰当的实验体系、流程和方法是实验成功的关键!
▲(上下滑动查看)▼
新品来袭:试用福利
诺唯赞长期助力表观遗传学研究,近期推出了 CUT&RUN(Vazyme #HD101、Vazyme #HD102)和 CUT&Tag(Vazyme #TD904)新品试剂盒,将于 2023 年 3 月 9 日 15:00 开展线上发布会,更有「新品体验官」前来分享实验经验和产品体验。
扫描下方二维码即可参加
小 V 期待与你的相会!
👇👇👇

新品活动 3 月清新来袭,活动详情请联系当地销售。更多产品信息可点击「阅读原文」或直接扫描下方二维码登录诺唯赞丁香商铺查询,免费领取试用装!
▼ 扫描二维码,免费申请试用 ▼


内容审核:潘成
项目审核:吴军
题图来源:图虫创意
参考文献:
[1] Skene, Peter J, and Steven Henikoff. eLife vol. 6 e21856. 16 Jan. 2017
[2] Wu, Shwu-Yuan et al. Molecular cell vol. 78,6 (2020): 1114-1132.e10.
[3]Kaya-Okur, Hatice S et al. Nature communications vol. 10,1 1930. 29 Apr. 2019
[4]Yin, Yuye et al. Nature immunology vol. 23,9 (2022): 1342-1354.
[5]Wu, Leiming et al. Journal of integrative plant biology vol. 64,11 (2022): 2033-2038.
点击「阅读原文」,免费申请试用