先导编辑(Prime editor, PEs)可以在没有供体DNA或双链断裂的情况下安装所需的碱基编辑,已经在植物中得到应用,原则上可以加速作物改良和育种。然而,它们在植物中的编辑效率普遍较低。根据熔体温度设计序列优化先导编辑向导RNA (prime editing guide RNA, pegRNA),使用双pegRNA和工程化PE均可提高PE效率。此外,基于水稻先导编辑实验数据,开发了pegRNA自动化设计平台PlantPegDesigner。
2022年11月25日,中国科学院遗传与发育生物学研究所高彩霞研究组在Nature Protocols 在线发表题为“Optimized prime editing in monocot plants using PlantPegDesigner and engineered plant prime editors (ePPEs)”的实验指南。该指南提出了使用PlantPegDesigner设计和优化pegRNAs的详细操作。
通过构建具有增强编辑效率的工程植物PE载体,用于启动编辑,使用报告系统评估编辑效率,并通过深度扩增子测序比较PEs的有效性和副产物。使用该方案,研究人员可以在4-7天内构建优化的pegRNAs进行先导编辑,并在3个月内获得先导编辑的水稻或小麦植株。
在植物基因组中引入所需的基因修饰在农业改良中起着重要作用。基于聚类规则间隔短回文重复序列(CRISPR)/Cas9的基因组编辑工具箱允许研究人员以灵活和精确的方式修改目标基因组DNA。先导编辑器(PEs)由工程Cas9 nickase (H840A)-逆转录酶(RT)融合蛋白和先导编辑向导RNA (pegRNA)组成,可用于对目标基因组位点产生改变,包括碱基替换、插入和删除。pegRNA包含引物结合位点(primer binding site, PBS)和RT模板(RT template, RTT)。Cas9 nickase (H840A)识别并切割靶位点的非靶DNA链,释放出单链DNA, PBS与释放的DNA杂交,作为反转录引物。然后,RTT编码的所需编辑被反向转录并转移到非目标DNA链上,生成一个DNA瓣,随后通过DNA修复将其合并到目标位点上。所有12种类型的点突变以及精确的插入和删除都可以通过先导编辑插入,而不需要双链断裂或供体DNA模板。尽管PEs能够对短DNA序列进行有针对性的修改,但它们不能轻易地产生大规模的插入或删除,或大规模的替换。然而,一些策略包括合并位点特异性重组酶和使用成对pegRNAs来克服这一问题,从而大大扩展了PEs的多功能性。该团队之前已经证明,PEs的效率受到pegRNA的设计的强烈影响。这里描述的造作流程是之前在水稻和小麦中植物启动基因组编辑方法的扩展。使用该方案,研究人员发现在水稻中,PEs的编辑效率与PBS序列的融化温度(melting temperature, Tm)密切相关,这可能是影响植物pegRNAs设计的主要因素。Tm为30°C的PBS序列在水稻中表现最佳。此外,在对正向链和反向链进行相同编辑的反式编码中使用两个pegRNAs也可以提高PE效率。
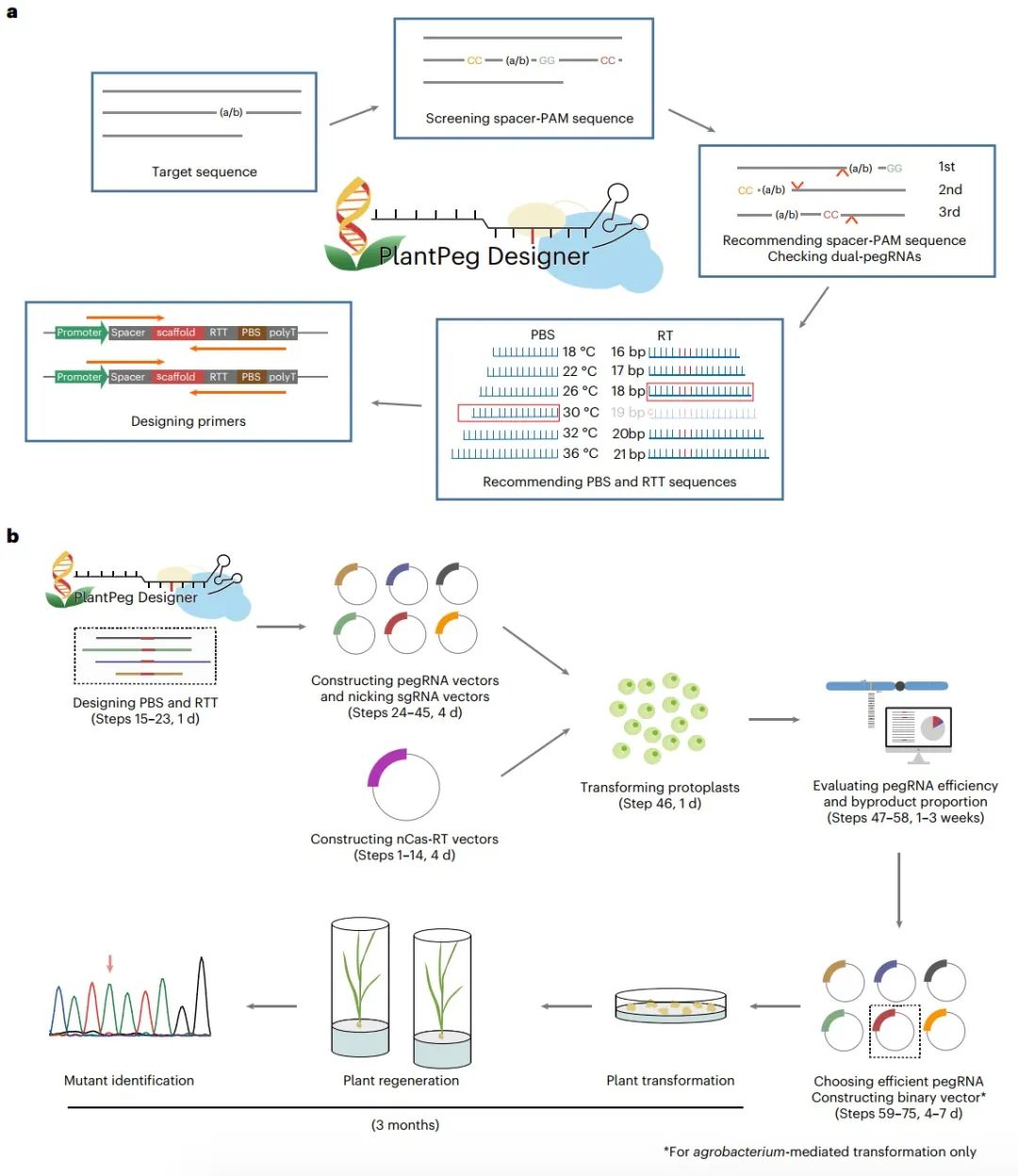 水稻和小麦的先导编辑流程(图源自Nature Protocols )结合这两种策略可以显著提高PE的效率。通过比较不同的PBS和RTT长度,以及所需编辑与切割位点之间的距离对水稻原生质体PE的影响,研究人员确定了水稻pegRNA设计的各种参数的最佳值。基于这些实验数据和上述策略,团队开发了一个web应用程序PlantPegDesigner,并通过实验验证了pegRNA设计算法。此外,研究人员还通过去除M-MLV (moloneymouine leukemia virus) RT的核糖核酸酶H结构域,结合具有核酸伴侣活性的病毒核衣壳蛋白,构建了工程植物素编辑器(engineered plant prime editor, ePPE),这提高了各种靶向修饰的频率,包括碱基替换,以及≤90 bp和34 bp插入的删除,平均5.8倍。通过将该方案中的pegRNA设计策略与ePPE相结合,在植物细胞中获得高效的先导编辑。综上所述,先导编辑提供了一种在真核细胞的目标位置插入各种编辑类型的新方法。虽然目前PlantPegDesigner推荐的最佳参数值是为水稻设置的,但通过测试和调整参数值,该工具可以适用于其他生物。高彩霞研究组博士后靳帅、林秋鹏为该论文的第一作者,高彩霞研究员为通讯作者。北京齐禾生科生物科技有限公司的高强博士也参与了此项工作。
水稻和小麦的先导编辑流程(图源自Nature Protocols )结合这两种策略可以显著提高PE的效率。通过比较不同的PBS和RTT长度,以及所需编辑与切割位点之间的距离对水稻原生质体PE的影响,研究人员确定了水稻pegRNA设计的各种参数的最佳值。基于这些实验数据和上述策略,团队开发了一个web应用程序PlantPegDesigner,并通过实验验证了pegRNA设计算法。此外,研究人员还通过去除M-MLV (moloneymouine leukemia virus) RT的核糖核酸酶H结构域,结合具有核酸伴侣活性的病毒核衣壳蛋白,构建了工程植物素编辑器(engineered plant prime editor, ePPE),这提高了各种靶向修饰的频率,包括碱基替换,以及≤90 bp和34 bp插入的删除,平均5.8倍。通过将该方案中的pegRNA设计策略与ePPE相结合,在植物细胞中获得高效的先导编辑。综上所述,先导编辑提供了一种在真核细胞的目标位置插入各种编辑类型的新方法。虽然目前PlantPegDesigner推荐的最佳参数值是为水稻设置的,但通过测试和调整参数值,该工具可以适用于其他生物。高彩霞研究组博士后靳帅、林秋鹏为该论文的第一作者,高彩霞研究员为通讯作者。北京齐禾生科生物科技有限公司的高强博士也参与了此项工作。原文链接:https://www.nature.com/articles/s41596-022-00773-9
